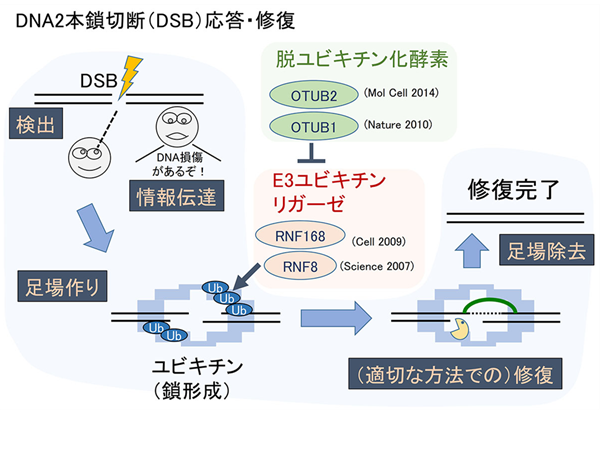
DNA修復は、
DNA損傷の発見、情報の伝達、DNA修復のための足場作り、DNA修復
そして、足場の除去という過程を経て
行われます。
修復の足場としては、
ユビキチンという小さなタンパクが鎖状に連結したものが使われます。
我々は(昔の研究室所属時も含めて)
足場を作る分子として
E3ユビキチンリガーゼRNF8,RNF168を
足場の作成を抑制する分子として
脱ユビキチン化酵素のOTUB1,OTUB2
を同定し、それらの分子の機能を解析する
研究に携わってきました。
ユビキチン関連のDNA損傷応答
Orchestration of the DNA-damage response by the RNF8 ubiquitin ligase
Science. 318:1637-40, 2007 (Durocher Lab)
E3 ubiquitin ligase RNF8とE2 ubiquitin conjugating enzyme UBC13がクロマチンをLysin 63-linkedユビキチン化(分解非誘導型ユビキチン化)することにより、DNA2本鎖損傷応答シグナルが細胞内で伝達されることを世界で初めて発見した。RNF8の発現が抑制されると、DNA損傷応答シグナルが完全に途絶し、細胞周期チェックポイントの機能不全が起こり、また、細胞の放射線抵抗性が著しく低下することを解明した。
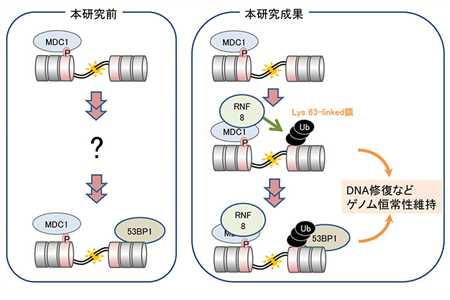
The RIDDLE syndrome protein mediates a ubiquitin-dependent signaling cascade at sites of DNA damage
Cell. 136(3):420-34, 2009 (Durocher Lab)
RNF8により作られたユビキチン鎖に別のE3 ubiquitin ligase RNF168 が結合することで、DNA損傷部位における様々な分子のユビキチン化が行われることを示した。また、RNF168は免疫不全を伴う疾患RIDDLEの責任遺伝子であることを示した。後の研究により、RNF168はヒストンH2Aをユビキチン化し、このユビキチン化H2Aに53BP1が結合することが示された。
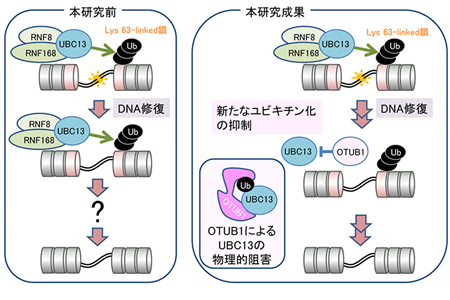
Non-canonical inhibition of DNA damage-dependent ubiquitination by OTUB1
Nature. 466: 941-6, 2010.
DNA2本鎖切断(DSB)応答シグナリングにおけるLys 63-linkedユビキチン化の抑制因子として、脱ユビキチン化酵素OTUB1を同定した。OTUB1は、Lys 63-linkedユビキチン鎖を切断するのではなく、ユビキチンがチャージしたE2酵素UBC13に結合して酵素活性非依存的に物理的にE2活性を阻害する(このような機構は全く知られていなかった)ことを解明した。OTUB1は、ユビキチン化依存性DNA損傷応答がDNA修復後に活性化しないように、E2酵素を抑制すると考えられる。
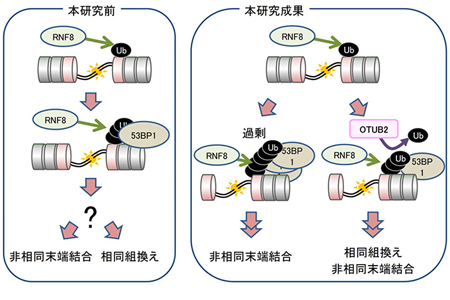
Fine-tuning of DNA damage-dependent ubiquitination by OTUB2 supports the DNA repair pathway choice
Mol Cell, 53, 4, 617-630, 2014.
脱ユビキチン化酵素OTUB2がRNF8によるユビキチン化にブレーキをかけることでDNA損傷応答の強さを適切に制御することを解明した。OTUB2によるユビキチン化制御が行われない場合、変異を伴いやすい非相同末端結合が誘導され、正確な修復経路である相同組換えが選択されにくくなる。その結果、細胞は、相同組換え修復を必要とするDNA損傷に対して高感受性となることが示された。
ユビキチン関連以外のDNA損傷応答
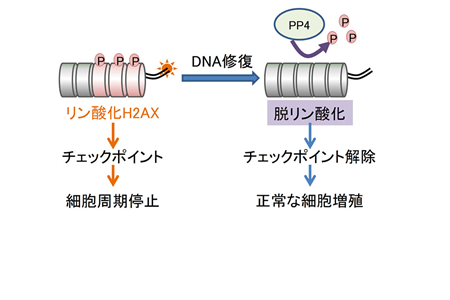
PP4 is a gamma H2AX phosphatase required for recovery from the DNA damage checkpoint
EMBO rep. 9:1019-26, 2008. (Durocher Lab)
DNA2本鎖切断(DSB)部位では、ヒストンH2AXがリン酸化されることで、DNA損傷応答の活性化が始まる。本研究では、DNA修復に伴い、(1)脱リン酸酵素PP4がリン酸化ヒストンH2AXを脱リン酸し、これにより(2)DNA損傷応答シグナルが停止し、(3)活性化されていた細胞周期チェックポイントが解除され、(4)細胞周期が正常に回復することを解明した。